Introdução
Células continuamente transitam entre diferentes estados funcionais através do desenvolvimento e da vida. Durante essa transição, a expressão gênica muda dinamicamente com a ativação de alguns genes, enquanto outros são silenciados. O sequenciamento de RNA de células únicas (scRNA-seq), permite aos pesquisadores identificar essa mudança dinâmica em alta resolução.
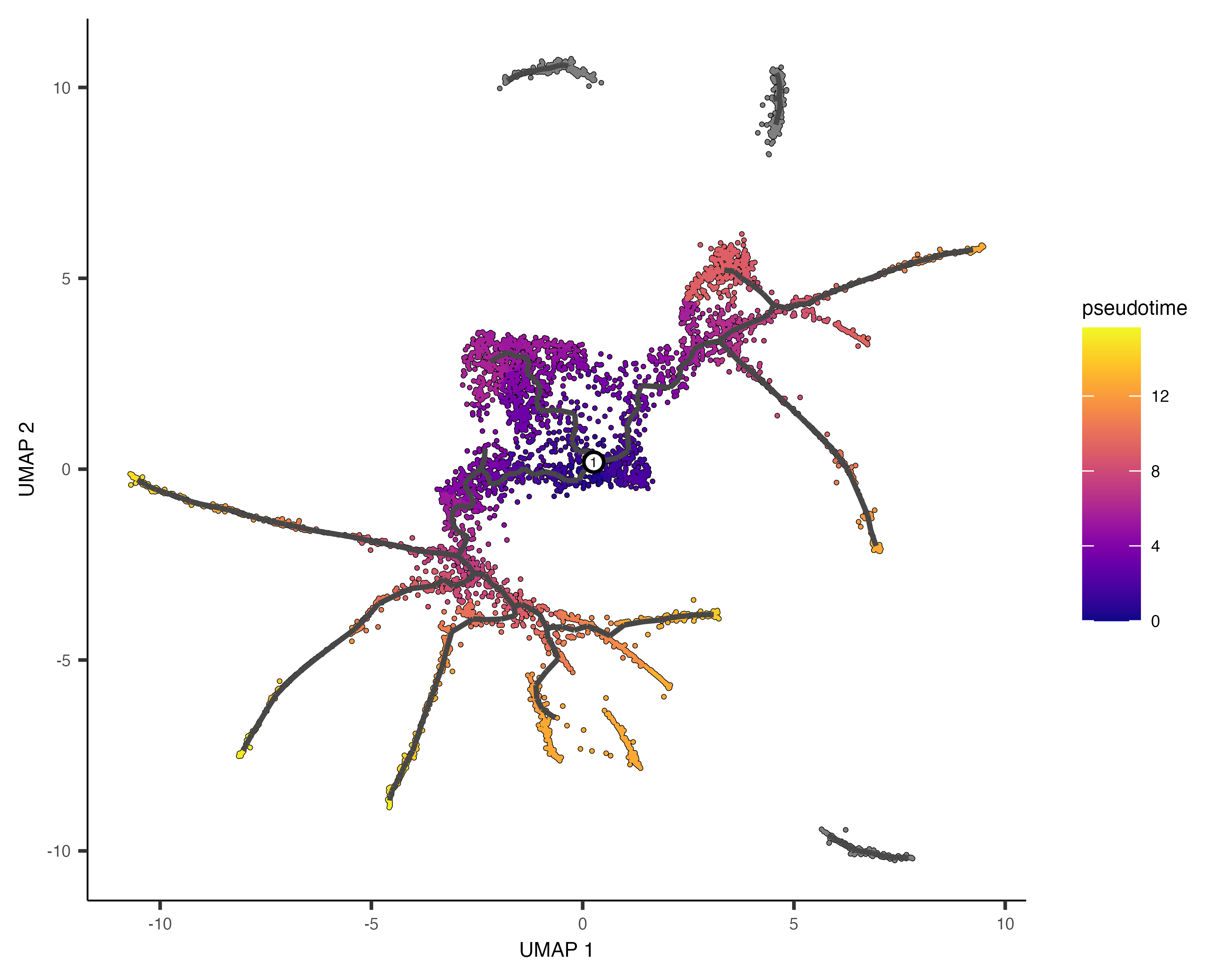
Ferramentas computacionais como o Monocle3 usa dados de scRNA-seq para reconstruir trajetórias celulares, nos ajudando a compreender como células progridem através de diferentes estados ao longo do tempo. Essa abordagem é particularmente útil em estudos de diferenciação, progressão de doenças e reprogramação celular.
Nesse tutorial, nós vamos aprender como inferir trajetórias celulares e estimar o pseudotempo - uma medida da progressão relativa das células ao longo do desenvolimento - usando o Monocle3. Ao analisar dados de células únicas, nós podemos mapear como células evoluem através de diferentes estados funcionais e identificar genes chave que direcionam essas transições.
Esse tutorial é inspirado e baseia-se em guias de estudos anteriores que demonstram o poder da inferência de trajetória na biologia de estudos de células únicas.
- Turorial original do Monocle3
- Tutorial combinando o Seurat e o Monocle3 pelo Stuart Lab
- Tutorial combinando Seurat and Monocle3 pela Mahima Bose
# Carregue as bibliotecas necessárias para as análises de sequenciamento de células únicas
library(monocle3) # Inferência de trajetórias
library(Seurat) # Estrutura de análises de células únicas
library(SeuratData) # Conjuntos de dados de células únicas pré-processados
library(SeuratWrappers) # Funcionalidades adicionais do Seurat
library(patchwork) # Composições de plotagens
library(harmony) # Correção de lotes
library(ggplot2) # Visualização da dados